2. 肇庆市教育局, 肇庆 526020
2. Zhaoqing Municipal Education Bureau, Zhaoqing 526020, China
结直肠癌, 是一种常见、致命的消化道恶性肿瘤, 其发病率和死亡率逐年上升且呈年轻化趋势. 结肠息肉易发生恶性病变, 诱发结直肠癌, 结肠镜检查能够有效筛查结肠息肉, 确定息肉位置以及外观信息, 帮助医生诊断, 实现早期治疗[1]. 然而, 临床实践中因息肉大小、形状、颜色、纹理各异, 只有经验丰富的医生才能准确诊断, 但受精力限制和情绪波动等影响, 诊断往往存在漏检或误检的情况. 为辅助诊断, 降低误诊概率, 研究一种能够准确分割结肠息肉的方法, 具有重要的临床意义与积极的应用前景.
结肠镜图像易受如光照、气泡、拍摄设备等因素影响, 出现模糊、伪影; 同时, 息肉外观各异, 边缘模糊, 分割难度大. 基于深度学习的医学图像分割方法发展至今, 涌现出各种模型结构针对各种医学场景与存在的问题提出了各种解决方法. Long等[2]提出的全卷积神经网络(FCN)最早将卷积神经网络应用于图像分割任务, 实现像素级别的分类, 由此揭开了语义级别医学图像分割研究的序幕. Ronneberger等[3]在2015年提出基于编码器-解码器结构的U-Net网络, 引入跳跃连接实现特征传递, 融合浅层与深层特征, 补充采样操作所损失的细节特征. 近年来, 研究人员围绕U-Net开展大量研究, 对网络结构进行诸多改进与扩展. 针对息肉分割场景, Fan等[4]提出的PraNet通过反向注意力模块建立分割区域与边界的联系, 增强分割性能; 为消除息肉颜色的影响, Zhong等[5]提出的SANet将图像内容与颜色解耦, 使模型更加关注息肉外观, 增强分割性能; 针对息肉定位不准确和边缘模糊的问题, Zhao等[6]提出的M2SNet通过具有不同感受野的金字塔减法单元捕捉多尺度息肉特征, 具有良好的分割精度和泛化能力.
2017年, Google提出应用于自然语言处理领域的Transformer[7], 研究人员开始将Transformer引入视觉任务中. ViT (vision Transformer)[8]首次将Transformer应用于图像分类任务, 达到超越ResNet[9]的精度, 具有更强的全局感知能力和适应性. 在医学图像分割领域, Chen等[10]提出的TransUNet首次将Transformer应用于U型网络结构, 其在编码器深层阶段引入Transformer建立长距离特征关系, 提高分割性能. 针对息肉分割场景, Zhang等[11]提出TransFuse, 通过并行结构结合应用多种注意力机制的BiFusion模块捕获多层次特征, 在多个数据集上验证了方法有效性; Wang等[12]提出由金字塔Transformer和多级特征聚合结构的渐进局部解码器构成的SSFormer, 提高模型对局部特征的处理能力.
结肠息肉分割方法发展至今取得显著进步, 但仍存在分割边缘模糊、分割效果有待进一步提高的问题. 现有的分割方法采取单分支网络结构, 其连续的下采样、上采样操作会造成部分细节信息丢失, 同时, 不同层次的特征信息无法或不便进行交互, 带来了分割边界模糊、分割效果不佳的问题. 针对这些问题, 提出基于局部-全局特征交互的双分支结肠息肉分割网络, 主要包括以下工作.
(1)针对单分支网络结构连续采样操作造成部分细节信息丢失和视野受限问题, 提出采用基于CNN的CSPDarknet53[13]网络与基于Transformer的PvTv2[14]网络的双分支网络结构, 逐层提取的息肉局部细节特征与全局语义特征, 扩大同层次特征视野, 提高对息肉边缘的判断与定位能力; 同时, 采用结合注意力门[15]的跳跃连接机制进一步突出边界信息, 增强边缘区域分割性能.
(2)为挖掘不同层次特征的依赖关系, 设计特征协同交互模块, 动态感知并聚合跨层次的特征交互信息, 实现不同层次特征信息的交互, 应对息肉病变区域外观各异的挑战; 同时, 为强化特征, 抑制背景噪声, 设计特征增强模块, 应用注意力机制强化息肉区域特征信息, 提高对息肉的精确识别与定位.
(3)相较于U-Net、PraNet、TransFuse、M2SNet等现有模型, 本文所提出的模型在Kvasir[16]、ClinicDB[17]、ColonDB[18]以及ETIS[19]这4个结肠息肉分割数据集上均取得更高的
大小、形状、颜色、纹理的多变性以及肠壁分界模糊给结肠息肉分割带来巨大挑战. 针对单分支网络连续采样操作导致部分细节信息丢失以及不同层次特征信息无法交互进而导致分割效果不佳的问题, 提出一种基于局部-全局特征交互的双分支结肠息肉分割网络, 模型总体结构如图1所示, 其编码阶段由局部特征提取模块(local feature extraction module, LFEM)、全局特征提取模块(global feature extraction module, GFEM), 特征协同交互模块(feature cooperative interaction module, FCIM)以及特征增强模块(feature enhancement module, FEM)组成, 解码阶段由上采样模块(upsample module, UM)组成.
编码阶段, 模型通过由CSPDarknet53构成的局部特征提取模块与PvTv2构成的全局特征提取模块并行提取多尺度的息肉局部细节特征与全局语义特征, 扩大同层次特征视野的同时减少因连续采样操作造成部分细节信息的丢失, 提高对息肉边缘的判断与定位能力; 随后, 为挖掘不同层次特征间的依赖关系, 设计特征协同交互模块, 应用哈达玛积、卷积与通道注意力, 结合深监督机制[20]动态感知并聚合跨层次的特征交互信息, 实现不同层次特征信息的交互, 应对息肉外观各异的挑战; 之后, 通过应用空间与通道注意力的特征增强模块强化病变区域特征, 抑制背景噪声, 提高对息肉的精确识别与定位; 同时, 应用结合注意力门(attention gate, AG)的跳跃连接机制进一步突出边界信息, 提高边缘区域的分割精度.
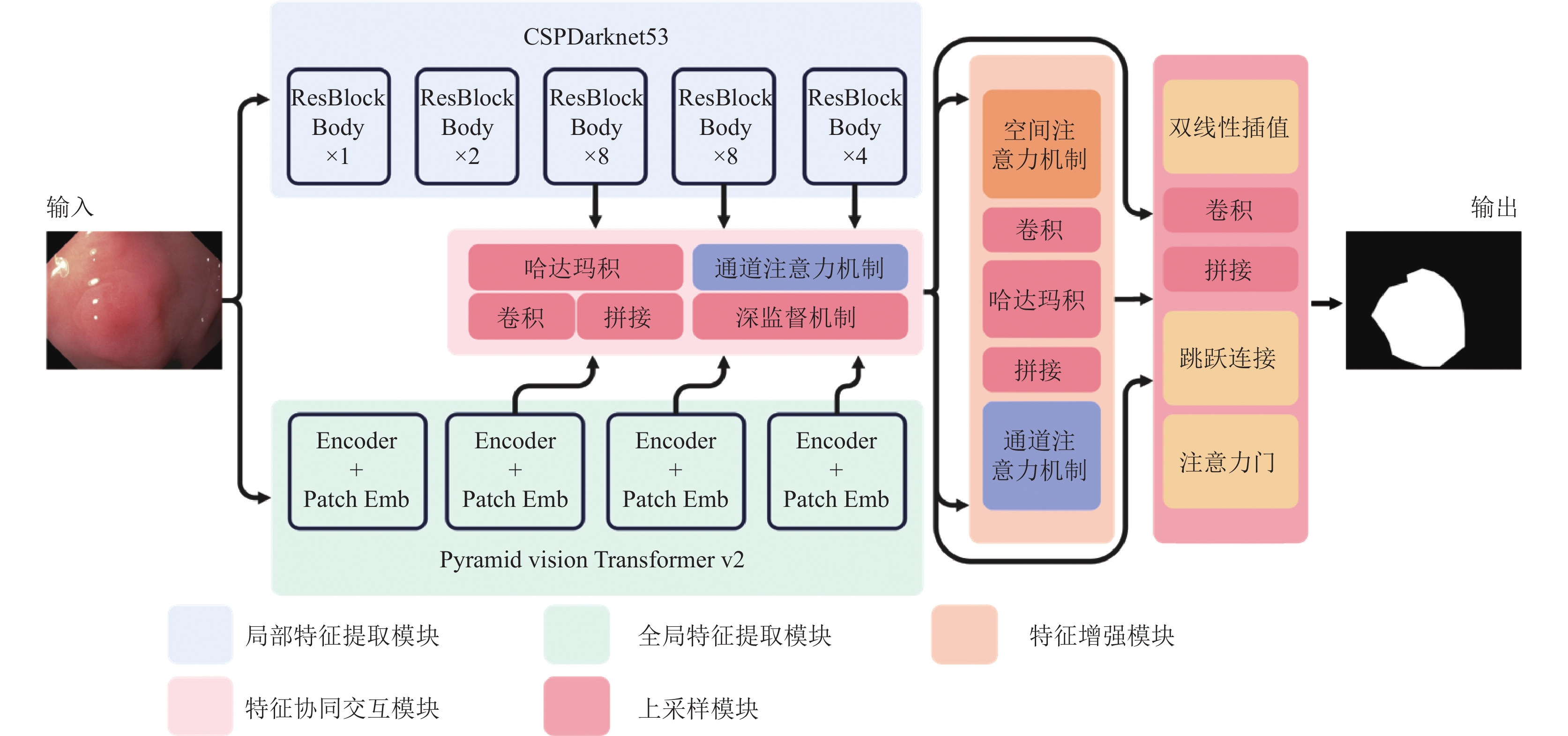
|
图 1 网络结构图 |
1.1 局部特征提取模块
CSPDarknet53构成的局部特征提取模块通过分层次特征重塑图像结构, 提取不同尺度下的局部细节特征. CSPDarknet53由应用
针对网络优化过程中因梯度重复导致的梯度消失问题, 残差模块体应用跨阶段合并思想, 将输入特征图经
全局特征提取模块(如图3)采用PvTv2 (pyramid vision Transformer v2)进行全局关系建模, 逐层提取全局语义特征, 扩大同层次特征视野. PvTv2包含4个阶段, 每个阶段均由嵌入层(Patch Emb)以及两个Transformer编码器(Transformer encoder)构成, 其在每一阶段应用渐进缩减策略缩小特征图分辨率以获得不同尺度的特征图. 具体来说, 在每一阶段, PvTv2首先将输入特征图分解为多个
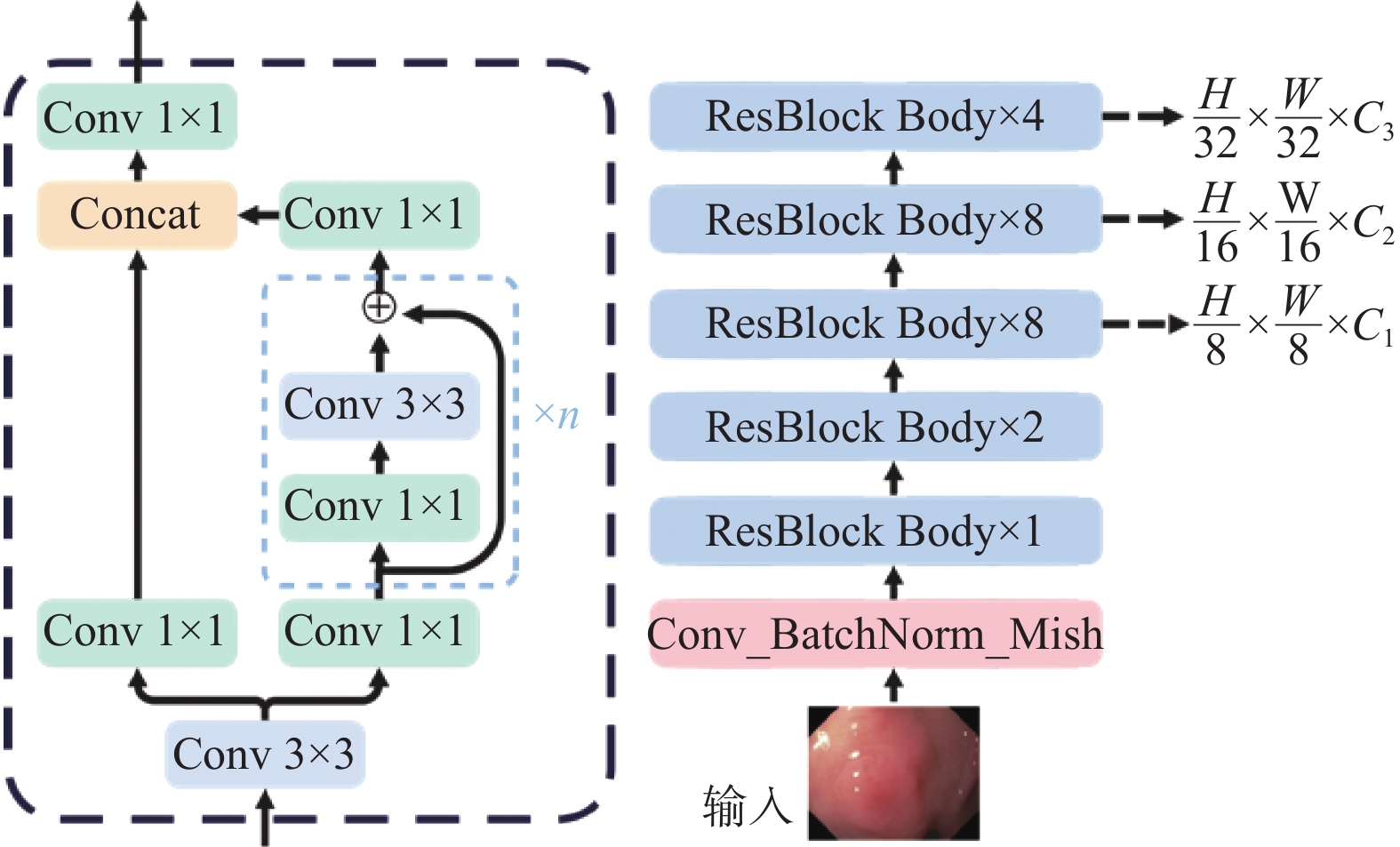
|
图 2 局部特征提取模块CSPDarknet53 |

|
图 3 全局特征提取模块 PvTv2 |
Transformer编码器由自注意力(self attention)层与结合深度可分离卷积的前馈网络(feed forward network)层组成, 深度可分离卷积包含深度卷积与逐点卷积, 分别对通道与空间上进行卷积操作, 能够避免特征混淆, 提高模型的计算效率; 同时, 自注意力层中的残差连接实现了同层次特征融合, 保留关键信息的同时减少因采用渐进缩减策略造成的部分信息丢失. 另外, 针对注意力机制存在的计算开销问题, PvTv2在Transformer编码器中提出空间缩减注意力机制替代原有的多头注意力(multi-head self attention)机制, 以
为充分利用不同层次、不同尺度特征的互补性, 利用深层语义特征对浅层细节特征的引导与增强, 如图4所示, 特征协同交互模块动态感知并聚合跨层次的特征交互信息, 实现不同层次特征信息的交互, 以应对息肉外观变化的挑战. 模块可分为3个阶段, 每阶段中来自局部与全局特征提取模块的特征具有相同的空间与通道维度. 具体来说, 利用哈达玛积与卷积对同层次的局部与全局特征进行交互, 利用特征间的互补性实现有效整合, 得到交互后的特征
为促进特征的充分交互, 引入深监督机制, 结合由加权损失和加权损失构成的联合损失函数, 对局部特征
如图4所示, 特征增强模块应用空间与通道注意力强化息肉区域病变区域特征, 抑制背景噪声, 提高对息肉的精确识别与定位. 具体来说, 应用SAM (spatial attention module)[25]对局部特征
| $ {{{L}}_{{c}}} = Concat(AvgPool({{L}}), MaxPool({{L}})) $ | (1) |
| $ {{{L}}_{{{{\mathrm{attn}}}}}} = {{L}}\times {\textit{Sigmoid}}(Conv({{{L}}_{{c}}})) $ | (2) |
如图5所示, SAM将局部特征

|
图 4 特征协同交互模块、特征增强模块、上采样模块 |
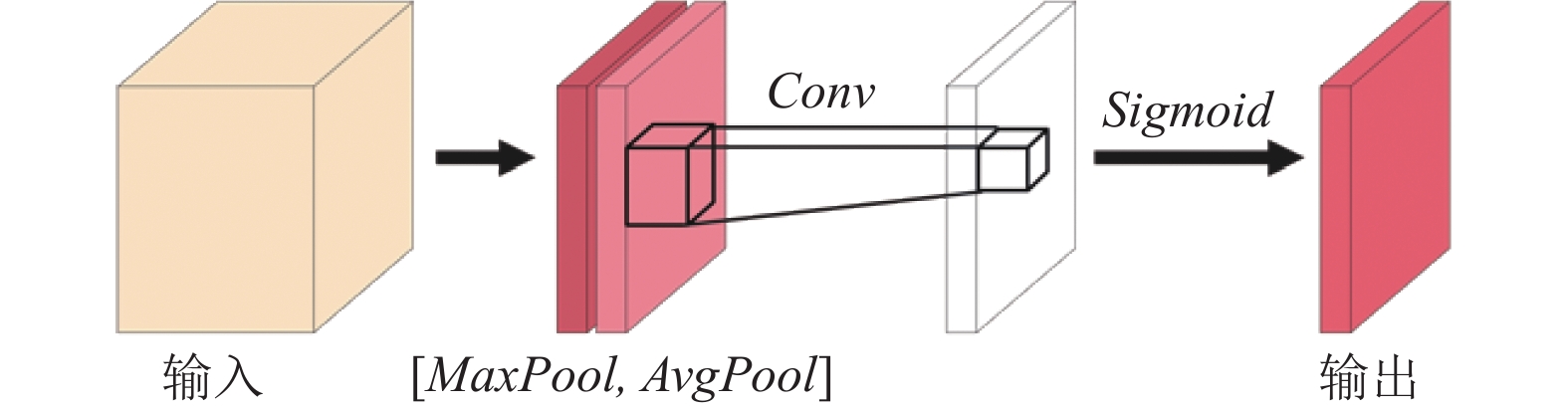
|
图 5 空间注意力机制 SAM |
针对全局特征
| $ {{{G}}_{{a}}} = {{GlobalAvgPool}}({{G}}) $ | (3) |
| $ {{{G}}_{{{attn}}}} = {{G}}\times{{\textit{Sigmoid}}} ({{Dense}}({{{G}}_{{a}}})) $ | (4) |
如图6所示(*表示逐通道乘法), SENet将全局特征
| $ {{Z}} = Conv(HadamardProduct({{L}}, {{G}})) $ | (5) |
| $ {{F}} = Conv(Concat({{{L}}_{{{{\mathrm{attn}}}}}}, {{{G}}_{{{{\mathrm{attn}}}}}}, {{Z}})) $ | (6) |
最后, 如式(5)与式(6), 应用哈达玛积Hadamard-Product与卷积将局部特征

|
图 6 通道注意力机制 SENet |
1.5 上采样模块
上采样模块应用结合注意力门的跳跃连接机制进一步突出边界信息, 提高边缘区域的分割精度. 如式(7)所示, 上采样模块将
| $ {{{W}}_i} = AG({\textit{Up}}({{F}}), {{{T}}_i}),{\text{ }}i = 1, 4 $ | (7) |
| $ {{F}} = Conv(Concat({\textit{Up}}({{F}}), {{{W}}_i})),{\text{ }}i = 1, 4 $ | (8) |
具体来说, 本模块采用双线性插值方法, 相比于反卷积、最近邻插值等方法, 其计算复杂度低, 能够保持分割图像的平滑性和连续性, 有助于恢复边缘细节. 注意力门的算法流程图如图7所示, 其将来自先前模块的特征
对于边缘信息要求高的息肉分割任务, 选择合适的损失函数是提高分割精度的关键. 传统
| $ Loss = Los{s_{IoU}} + Los{s_{BCE}} $ | (9) |

|
图 7 注意力门 AG |
具体来说, 本文将加权
| $ Los{s_{IoU}} = 1 - \frac{{\displaystyle\sum\limits_{i = 1}^N {{w_i} \times {y_i} \times {{\hat y}_i}} }}{{\displaystyle\sum\limits_{i = 1}^{{N}} {{w_i} \times ({y_i} + {{\hat y}_i} - {y_i} \times {{\hat y}_i})} }} $ | (10) |
其中,
加权损失
| $ Los{s_{BCE}} = - \frac{1}{N}\sum\limits_{i = 1}^N {{w_i}} \times [{y_i}\log ({\hat y_i}) + (1 - {y_i})\log (1 - {\hat y_i})] $ | (11) |
其中,
本文配置的实验环境如下: CPU Intel i9 11900K/F, 内存 64 GB, GPU NVIDIA GeForce RTX 3090, 显存 24 GB, Python 3.8, Linux Ubuntu 操作系统, PyTorch深度学习框架.
2.1 实验数据集实验采用胃肠道疾病研究的医学图像公开数据集Kvasir、ClinicDB、ColonDB以及ETIS. Kvasir数据集共包含1000张胃肠道息肉图片及其对应的分割标签图, 涵盖息肉、溃疡、出血等症状且存在检查中易被忽略的直径小于10 mm的息肉; ClinicDB与ColonDB数据集分别包含612张与380张图片及其对应的分割标签图; ETIS数据集包含来自不同地区医疗中心的不同内镜设备中提取出来196张结肠息肉图片及其对应的分割标签图. 具体来说, 训练数据集由来自Kvasir数据集的900张图像及ClinicDB数据集的550张图像组成, 共包含1450个样本及其对应的分割标签图; 测试数据集共由来自Kvasir数据集的100张图像, Clinic-DB数据集的62张图像, ColonDB数据集的380张图像以及ETIS数据集的196张图像组成, 共包含738个样本及其对应的分割标签图; 消融实验测试数据集由来自ETIS数据集的196张图像及其对应的分割标签图组成.
如图8所示, 实验所使用的数据集在息肉大小、形状、纹理上存在多变性, 且肠壁分界模糊, 同时成像质量存在不稳定性, 受到如光照、气泡、拍摄条件等因素影响, 实际分割存在巨大挑战.
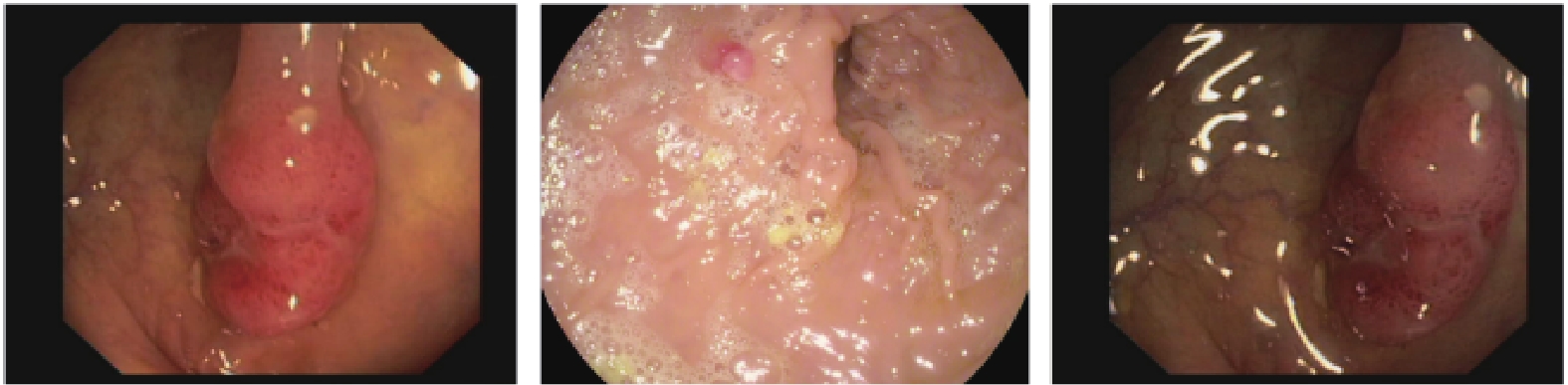
|
图 8 数据集图片 |
2.2 实验参数与评价指标
实验参数设置如下: 初始学习率为1E–4, 衰减率为0.1, 衰减周期为30轮次, 使用AdamW[26]优化器, 共进行100轮次训练; 数据增强方法采用归一化与0.75倍、1倍、1.25倍的多尺度输入方法.
实验选取
| $ Dice = \frac{{2|A \cap B|}}{{|A| + |B|}} $ | (12) |
其中,
| $ mDice = \frac{1}{n}(Dic{e_1} + Dic{e_2} + \cdots + Dic{e_n}) $ | (13) |
| $ IoU = \frac{{A \cap B}}{{A \cup B}} $ | (14) |
其中,
| $ mIoU = \frac{1}{n}(Io{U_1} + Io{U_2} + \cdots + Io{U_n}) $ | (15) |
为验证模型的有效性与鲁棒性, 在使用相同测试数据集的基础上, 选取一系列模型开展对比实验.
如表1所示, 相较于U-Net, PraNet等CNN网络模型, 结合CNN与Transformer的TransFuse, 采用不确定性增强的上下文注意力机制的UACANet以及多尺度减法网络M2SNet等, 本文提出的模型在4个测试数据集上均取得更高
| 表 1 不同网络在Kvasir, ClinicDB, ColonDB, ETIS数据集上的对比实验结果 |
|
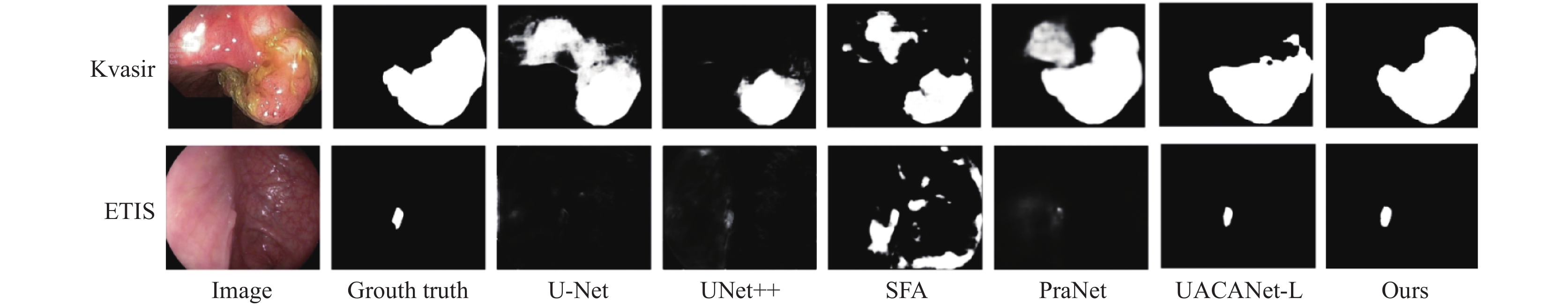
图 9 部分模型在Kvasir、ETIS数据集上的分割结果 |
2.4 消融实验结果与分析
为验证本文所提出的模块、方法对模型性能提升的有效性和必要性, 基于相同的实验环境、实验参数以及训练数据集, 开展相应的消融实验. 实验中, 本文将表1中所提出网络的分割指标作为比较基准, 分别进行6项消融实验, 使用一致的评价指标
| 表 2 网络在ETIS数据集上的消融实验结果 |
第1项消融实验单纯去除特征协同交互模块中的深监督机制, 网络在
息肉边缘的准确分割对于分割性能的提升十分重要. 第2项消融实验去除结合注意门的跳跃连接机制, 网络在
第3项消融实验去除特征增强模块, 网络在
第4项消融实验去除结合注意门的跳跃连接机制以及特征协同交互模块, 网络在
第5项消融实验去除特征增强模块与结合注意力门的跳跃连接机制, 第6项消融实验在第5项实验的基础上去除特征协同交互模块. 相比前4项实验, 网络在与指标上均出现了更大程度的下降, 由此可见, 以上模块的有机结合十分重要; 具体而言, 特征协同交互模块动态感知并聚合跨层次的特征交互信息, 为后续特征增强模块提供更为丰富、适应息肉外观变化的特征信息, 并应用注意力机制进一步强化病变区域特征, 抑制背景噪声; 同时与结合注意力门的跳跃连接机制进一步细化边缘特征, 显著提升网络的分割性能与泛化性能.
3 结论与展望大小、形状、颜色、纹理的多变性以及肠壁分界模糊给结肠息肉分割带来巨大挑战. 针对单分支网络连续采样操作导致部分细节信息丢失以及不同层次特征信息无法交互进而导致分割效果不佳的问题, 本文提出一种基于局部-全局特征交互的双分支结肠息肉分割网络. 网络在特征提取阶段构建并行网络结构, 使用CSPDarknet53网络与PvTv2网络逐层捕获息肉局部细节特征与全局语义特征, 扩大同层次特征视野的同时减少因连续采样操作造成的部分细节信息丢失, 提高对息肉边缘的判断与定位能力; 为充分利用不同层级、不同尺度特征信息的互补性, 利用深层语义特征对浅层细节特征的引导与增强, 提出特征协同交互模块动态感知并聚合跨层次交互信息, 实现不同层次特征信息的交互, 以适应息肉外观变化的挑战; 设计特征增强模块, 应用空间与通道注意力机制强化息肉区域特征, 抑制背景噪声, 提高对息肉的精确识别与定位; 最后, 采用结合注意力门的跳跃连接机制进一步突出边缘信息, 增强边缘区域分割性能. 网络在ClinicDB、Kvasir、ColonDB、ETIS这4个息肉分割数据集上与主流基线网络进行对比实验以验证有效性; 同时, 在ETIS数据集上进行消融实验以验证所提出模块的有效性; 结果表明, 本文所提出网络能够有效提升息肉分割精度, 在临床诊断中具有一定的辅助应用价值, 但仍存在不足, 后续研究中将改进各个模块, 加强对小目标、低对比度场景下的息肉检测能力, 进一步提高分割准确性与稳定性.
| [1] |
Zauber AG, Winawer SJ, O’Brien MJ, et al. Colonoscopic polypectomy and long-term prevention of colorectal-cancer deaths. The New England Journal of Medicine, 2012, 366(8): 687-696. DOI:10.1056/NEJMoa1100370 |
| [2] |
Long J, Shelhamer E, Darrell T. Fully convolutional networks for semantic segmentation. Proceedings of the 2015 IEEE Conference on Computer Vision and Pattern Recognition. Boston: IEEE, 2015. 3431–3440.
|
| [3] |
Ronneberger O, Fischer P, Brox T. U-Net: Convolutional networks for biomedical image segmentation. Proceedings of the 18th International Conference on Medical Image Computing and Computer-assisted Intervention. Munich: Springer, 2015. 234–241.
|
| [4] |
Fan DP, Ji GP, Zhou T, et al. PraNet: Parallel reverse attention network for polyp segmentation. Proceedings of the 23rd International Conference on Medical Image Computing and Computer Assisted Intervention. Lima: Springer, 2020. 263–273.
|
| [5] |
Zhong ZL, Lin ZQ, Bidart R, et al. Squeeze-and-attention networks for semantic segmentation. Proceedings of the 2020 IEEE/CVF Conference on Computer Vision and Pattern Recognition. Seattle: IEEE, 2020. 13062–13071.
|
| [6] |
Zhao XQ, Jia HP, Pang YW, et al. M2SNet: Multi-scale in multi-scale subtraction network for medical image segmentation. arXiv:2303.10894, 2023.
|
| [7] |
Vaswani A, Shazeer N, Parmar N, et al. Attention is all you need. Proceedings of the 31st International Conference on Neural Information Processing Systems. Long Beach: ACM, 2017. 6000–6010.
|
| [8] |
Dosovitskiy A, Beyer L, Kolesnikov A, et al. An image is worth 16x16 words: Transformers for image recognition at scale. Proceedings of the 9th International Conference on Learning Representations. ICLR, 2021.
|
| [9] |
He KM, Zhang XY, Ren SQ, et al. Deep residual learning for image recognition. Proceedings of the 2016 IEEE Conference on Computer Vision and Pattern Recognition. Las Vegas: IEEE, 2016. 770–778.
|
| [10] |
Chen JN, Lu YY, Yu QH, et al. TransUNet: Transformers make strong encoders for medical image segmentation. arXiv:2102.04306, 2021.
|
| [11] |
Zhang YD, Liu HY, Hu Q. TransFuse: Fusing transformers and CNNs for medical image segmentation. Proceedings of the 24th International Conference on Medical Image Computing and Computer Assisted Intervention. Strasbourg: Springer, 2021. 14–24.
|
| [12] |
Wang JF, Huang QM, Tang FL, et al. Stepwise feature fusion: Local guides global. Proceedings of the 25th International Conference on Medical Image Computing and Computer Assisted Intervention. Singapore: Springer, 2022. 110–120.
|
| [13] |
Bochkovskiy A, Wang CY, Liao HYM. YOLOv4: Optimal speed and accuracy of object detection. arXiv:2004.10934, 2020.
|
| [14] |
Wang WH, Xie EZ, Li X, et al. PVT v2: Improved baselines with pyramid vision Transformer. Computational Visual Media, 2022, 8(3): 415-424. DOI:10.1007/s41095-022-0274-8 |
| [15] |
Schlemper J, Oktay O, Schaap M, et al. Attention gated networks: Learning to leverage salient regions in medical images. Medical Image Analysis, 2019, 53: 197-207. DOI:10.1016/j.media.2019.01.012 |
| [16] |
Jha D, Smedsrud PH, Riegler MA, et al. Kvasir-SEG: A segmented polyp dataset. Proceedings of the 26th International Conference on MultiMedia Modeling. Daejeon: Springer, 2020. 451–462.
|
| [17] |
Bernal J, Sánchez FJ, Fernández-Esparrach G, et al. WM-DOVA maps for accurate polyp highlighting in colonoscopy: Validation vs. saliency maps from physicians. Computerized Medical Imaging and Graphics, 2015, 43: 99-111. DOI:10.1016/j.compmedimag.2015.02.007 |
| [18] |
Tajbakhsh N, Gurudu SR, Liang JM. Automated polyp detection in colonoscopy videos using shape and context information. IEEE Transactions on Medical Imaging, 2016, 35(2): 630-644. DOI:10.1109/TMI.2015.2487997 |
| [19] |
Silva J, Histace A, Romain O, et al. Toward embedded detection of polyps in WCE images for early diagnosis of colorectal cancer. International Journal of Computer Assisted Radiology and Surgery, 2014, 9(2): 283-293. DOI:10.1007/s11548-013-0926-3 |
| [20] |
Lee CY, Xie SN, Gallagher PW, et al. Deeply-supervised nets. Proceedings of the 18th International Conference on Artificial Intelligence and Statistics. San Diego: AISTATS, 2015. 562–570.
|
| [21] |
Misra D. Mish: A Self Regularized non-monotonic activation function. Proceedings of the 31st British Machine Vision Conference. BMVC, 2020.
|
| [22] |
Rumelhart DE, Hinton GE, Williams RJ. Learning representations by back-propagating errors. Nature, 1986, 323(6088): 533-536. DOI:10.1038/323533a0 |
| [23] |
Glorot X, Bordes A, Bengio Y. Deep sparse rectifier neural networks. Proceedings of the 14th International Conference on Artificial Intelligence and Statistics. Fort Lauderdale: AISTATS, 2011. 315–323.
|
| [24] |
Hu J, Shen L, Sun G. Squeeze-and-excitation networks. Proceedings of the 2018 IEEE/CVF Conference on Computer Vision and Pattern Recognition. Salt Lake City: IEEE, 2018. 7132–7141.
|
| [25] |
Woo S, Park J, Lee JY, et al. CBAM: Convolutional block attention module. Proceedings of the 15th European Conference on Computer Vision (ECCV). Munich: Springer, 2018. 3–19.
|
| [26] |
Loshchilov I, Hutter F. Decoupled weight decay regularization. Proceedings of the 7th International Conference on Learning Representations. New Orleans: ICLR, 2019.
|
| [27] |
Zhou ZW, Rahman Siddiquee MM, Tajbakhsh N, et al. UNet++: A nested U-Net architecture for medical image segmentation. Proceedings of the 4th International Workshop on Deep Learning in Medical Image Analysis and Multimodal Learning for Clinical Decision Support. Granada: Springer, 2018. 3–11.
|
| [28] |
Fang YQ, Chen C, Yuan YX, et al. Selective feature aggregation network with area-boundary constraints for polyp segmentation. Proceedings of the 22nd International Conference on Medical Image Computing and Computer Assisted Intervention. Shenzhen: Springer, 2019. 302–310.
|
| [29] |
Patel K, Bur AM, Wang GH. Enhanced U-Net: A feature enhancement network for polyp segmentation. Proceedings of the 18th Conference on Robots and Vision (CRV). Burnaby: IEEE, 2021. 181–188.
|
| [30] |
Kim T, Lee H, Kim D. UACANet: Uncertainty augmented context attention for polyp segmentation. Proceedings of the 29th ACM International Conference on Multimedia. ACM, 2021. 2167–2175.
|
| [31] |
Nguyen M, Bui TT, Van Nguyen Q, et al. LAPFormer: A light and accurate polyp segmentation Transformer. arXiv: 2210.04393, 2022.
|
 2024, Vol. 33
2024, Vol. 33


