2. 山东浪潮优派科技教育有限公司, 济南 250101
2. Shandong Inspur-UPTEC Education Ltd., Jinan 250101, China
肝癌是全球发病率第6位和死亡率第4位的恶性肿瘤[1]. 最新数据显示, 中国肝癌年新发病例38.9万, 居恶性肿瘤第4位, 其年死亡病例为33.6万, 居恶性肿瘤死亡第2位, 疾病负担沉重[2]. 一般情况下, 如果影像学检查(CT, MRI)提示可能存在肝癌, 医生会通过肝穿刺活检、经皮肝穿刺活检等方法, 从患者的肝脏中取出一小块组织进行检查, 通过对肝肿瘤组织进行显微镜下的观察和分析, 可以确定是否存在癌细胞, 并确定癌细胞的类型、分期等信息. 而且, 一些病理特征还可以为临床治疗和预后提供指导, 如微血管侵犯(MVI)和微卫星结节. 然而, 目前肝癌的病理诊断实践是耗时的, 而且病理学家的短缺日益严重, 但由于病例数量增加, 以及需要更广泛的诊断来确定患者的最佳治疗方法, 他们的工作量也在增加[3]. 近些年来, 用于计算机辅助图像分析的深度学习技术[4]的出现允许通过训练卷积神经网络(CNN)在低到高级别上自动提取特征. 这种方法不需要手动特征设计, 降低了病理学家的工作量, 并且经过训练的模型可以几乎完美地识别对象[5].
CNN是一种深度学习模型, 主要由卷积层、池化层和全连接层组成. CNN最初是由LeCun等人[6]于1980年代末提出并应用于手写数字识别任务, 随着不断发展, CNN的架构也在不断优化和改进, 在很多领域得到了广泛应用. 在医学图像诊断上, CNN可以通过学习医学图像中的特征, 自动识别和分类病理区域, 从而帮助医生进行诊断和治疗. 文献[7]的研究结果证明, CNN对癌症图像分类的准确性达到了最佳水平. 此外, CNN还可以用于预测患者的疾病风险和预后.
对于肝癌诊断, 文献[8–13]是近期发表的几篇优秀的综述, 但是都未对病理学图像上使用CNN对肝癌诊断方法进行系统概述. 为了给肝癌辅助诊断相关研究领域的未来工作提供参考, 本文主要介绍了近几年CNN架构在病理图像中对肝癌诊断的应用, 其中包括肝肿瘤检测、图像分割以及术前预测这3个方面, 对每一方面, 都结合该领域应用成功的几种CNN架构进行讨论. 通过研究CNN 在病理图像上的多方面应用, 理清CNN关于肝癌的诊断算法, 指出目前存在的问题, 并对未来的发展趋势进行展望.
1 数据集介绍及评价标准使用CNN模型进行肝癌病理学图像诊断方法时, 数据集具有重要的作用. 本节介绍了几个常用的肝癌病理图像数据集以及相关的评价标准.
1.1 PAIP-2019 challengePAIP-2019 challenge数据集是一个用于肝脏病理图像分割的公开数据集, 病理数据取自于2005年–2018年6月在SNUH, SNUBH, SMG-SNUBMC采样的肝脏肿瘤组织[14]. 数据集包含100个全切片图像, 用于训练(50)、验证(10)和测试(40). 用于训练和验证的WSI具有两层对活肿瘤区域和整个肿瘤区域的注释.
1.2 TCGA-LIHCTCGA-LIHC是TCGA (The Cancer Genome Atlas)计划中的一个子项目, 专门研究肝细胞癌(hepatocellular carcinoma, HCC)的基因组学和转录组学特征. TCGA-LIHC数据集包括了超过300个肝细胞癌样本的临床和分子数据, 其中包括基因组测序、外显子组测序、RNA测序、DNA甲基化、蛋白质组学等多种高通量分子数据. 此外, 该数据集还提供了大量的样本描述信息和临床数据, 包括患者基本信息、病理学特征、治疗方案和治疗效果等[15].
1.3 KMC dataKMC肝脏数据集是一组用于肝癌诊断的医学图像数据集, 数据集包括来自不同患者的257张原始幻灯片, 每张大小为1920×1440, 属于4种不同亚型的肝HCC肿瘤. 每个像素的大小为3.69 μm×3.69 μm. 该数据集已由Manipal的KMC临床病理学家进行注释. 这个数据集可用于机器学习和深度学习模型的训练和评估, 以帮助医生自动识别和分类肝细胞癌肿瘤.
1.4 评价标准卷积神经网络在肝癌病理学图像诊断中的评价标准在每个阶段各有不同, 常用的评价指标如表1所示.
TP (真阳性): 表示模型正确地将正例样本预测为正例的数量. 在像素级别的分类任务中, 它表示模型正确地将正例像素(例如肿瘤区域)预测为正例. FP (假阳性): 表示模型错误地将负例样本预测为正例的数量. 在像素级别的分类任务中, 它表示模型错误地将负例像素(例如非肿瘤区域)预测为正例. FN (假阴性): 表示模型错误地将正例样本预测为负例的数量. 在像素级别的分类任务中, 它表示模型错误地将正例像素(例如肿瘤区域)预测为负例. TN (真阴性): 表示模型正确地将负例样本预测为负例的数量. 在像素级别的分类任务中, 它表示模型正确地将负例像素(例如非肿瘤区域)预测为负例.
| 表 1 常用评价指标 |
2 传统的机器学习算法正在肝癌诊断中的应用分析
在深度学习兴起之前, 肝癌的诊断主要依赖于传统的机器学习方法, 机器学习是一种利用数学和统计模型来构建算法, 使计算机能够从数据中学习和进行预测的方法. 其核心思想是从给定的训练数据中学习模型, 并将该模型应用于新的未知数据进行预测或分类, 在进行肝脏图像的诊断时, 首先需要手动设计和提取特征, 然后根据任务的需求选择适当的机器学习模型, 常用的手工特征提取的方法主要包括纹理特征提取、形状特征提取、颜色特征提取等. 需要注意的是, 手工特征提取需要根据具体的任务和应用场景进行选择和设计, 不同的特征提取方法可能适用于不同类型的图像和不同的应用场景. 文献[16–18]展示了使用机器学习算法进行肝癌诊断的实例, 如支持向量机(SVM)[19]、朴素贝叶斯分类器等.
机器学习方法广泛用于分析患者的临床信息、基因记录和医学图像. 证明了数据挖掘和机器学习工具在医学领域的巨大潜力. 这些工具可以从医学数据集中发现隐藏的重要预测参数, 从而提供疾病的早期预测和诊断[20]. 尽管这些方法在某些情况下可以取得令人满意的结果, 但它们往往受到手工特征提取和固定阈值的局限性, 无法充分利用图像中的信息. 而且, 手工设计和提取特征需要具备领域专业知识和经验, 并且对于复杂的肝脏图像数据来说, 特征的选择和设计并不是一项简单的任务. 随着机器学习技术的发展, 特别是深度学习方法的兴起, 越来越多的研究人员开始使用基于深度学习的方法进行图像分析. 相较于传统的机器学习方法, 基于深度学习的方法可以自动学习特征, 避免了手工特征提取的局限性, 而且可以通过端到端的学习从原始数据到最终结果, 充分利用图像中的信息, 提高肝癌图像分析的准确性和可靠性.
3 CNN在肝癌诊断中的应用分析随着数字化医疗和计算机图像处理技术的应用与发展, WSI随之出现. WSI的全称是全扫描数字病理图像, 是一种将病理学切片数字化的技术, 可以将整张病理学切片转化为高分辨率的数字图像[21]. 临床实践中, 活检切片可以与WSI相结合, 从而获得更全面、准确的肿瘤组织形态和结构信息, 图1是展示了带标注的肝肿瘤WSI图像示例. 为帮助医生更准确、高效地进行肝癌诊断, 研究人员利用 CNN 设计了肝肿瘤检测、图像分割、术前预测算法, 将结合具体网络架构进行探讨.

|
图 1 带标注的肝肿瘤WSI图像 |
3.1 肝肿瘤检测
检测肝肿瘤是肝脏病理图像分析的重要步骤之一. 最初阶段快速准确地检测, 可以获得治疗效果的好处最大化[22]. 传统的组织病理学检测方法由于工作量大, 给临床诊断带来了很大的负担[23]. 随着研究人员不断的深入研究, 尝试将卷积神经网络与病理图像相结合, 提出了很多提高肝癌肿瘤检测效率的新方法.
肿瘤细胞具有高度可变的形态和生长模, 在进行检测实验时, 只关注从单尺度的高倍斑块中提取的细胞水平的特征可能会失去对全局的宏观特征, 而且当发生变化的细胞区域被覆盖时, 周围细胞结构信息太少可能会导致检测失败. Huang等人[24]将多尺度CNN(MSCN)架构应用于肝细胞癌H&E染色组织学WSI中, 使用标准VGG16[25]的卷积层构建两个并行CNN提取多尺度特征来参考细胞水平和细胞结构信息, 实验结果显示, 该网络在HCC肿瘤检测任务中实现了91%的mIoU, 相比于单尺度CNN (SSCN)在实验中可以提供更好的性能. Yang等人[26]同样使用MSCN进行肝肿瘤检测实验, 并尝试在MSCN架构的基础上加入对齐模块和特征融合模块, 与单尺度卷积神经网络相比, MSCN提高了检测性能. 并且, 在MSCN基础上增加对齐模块和特征融合模块的方式, 实验效果优于单纯的MSCN模型.
特征信息的处理对实验结果会产生一定的影响, 局部区域的处理和特征提取关乎实验的准确性. Diao等人[27]在肝肿瘤检测实验中在数据预处理阶段尝试添加人工噪声图像作为补丁, 向我们展示了人工智能驱动的HCC病理图像诊断的关键问题, 即噪声注释. Hou等人[28]在进行肝肿瘤分类实验时使用基于补丁的卷积神经网络(CNN)模型, 将太大的WSI图像裁剪成各种补丁, 在实验时将这些补丁独立馈送至模型中, 该模型在全玻片组织图像(WSI)分类中取得了不错的结果, 证明了在小图像上, 使用基于补丁的CNN可以优于基于图像的CNN. 借鉴于其思想. Wang等人[29]在肝肿瘤检测实验中将PAIP-2019 challenge中的60个图像作为数据集, 使用基于补丁的卷积神经网络将肝肿瘤WSI中提取的补丁进行类别预测, 实验过程中除了对补丁集进行训练外还做了扩展数据集和细化分类效果, 为了获取更好的实验结果, 实验还加入了迁移学习, 有效地减少大图像处理的计算量和内存占用, 提高了肝肿瘤检测性能. 唯一遗憾的是在尝试使用多种数据增强技术进行组合时, 没有达到理想的精度.
实验中样本数据较少、数据注释量严重稀缺是肝肿瘤病理学图像检测实验最常见的问题, 缺乏足够的样本数据和注释信息会使模型很难从中学习到充分的肿瘤特征, 容易出现类不平衡和过拟合等现象, 极大限制了模型训练和性能评估的准确性和可靠性. 研究人员在实验过程中对于出现的这类问题提出了各自的思路和方法, 对于问题的解决取得了显著的成果. 卷积神经网络在肝肿瘤检测中的应用已经取得了很大的进展. 卷积神经网络和病理WSI图像的结合使得肝肿瘤检测更加的准确、高效. 值得注意的是, 在进行实验探究时, 还需要结合具体的临床应用需求和实际情况, 进行优化和改进, 以提高检测的可靠性和实用性. 卷积神经网络在肝肿瘤检测中的应用前景非常可期, 未来还有很大的发展空间.
3.2 病理图像分割肝癌病理学图像分割的实验研究最常见的是对肝细胞的分割和对肿瘤区域的分割. 肝细胞等结构的分割通常是为了确定肝癌细胞的位置和形态, 从而更好地评估肝癌的严重程度和预后工作. 而肿瘤区域分割则旨在确定肝癌的边界和大小, 以便更好地进行治疗规划和疾病监测. 两种方式可以互相补充, 共同提高肝癌图像分析的准确性和可靠性[30]. 这两种分割方式的分割方法都可分为无监督方法和监督方法. 无监督方法是指在训练过程中不需要标注数据, 通过对图像的特征进行聚类或分割, 例如Roy等人[31]. 但这类方法难以控制分割结果, 而且对数据质量要求高. 相比于无监督方法, 现有的研究中有监督方法具有更高的准确性、更好的可控性和更广泛的适用性.
有监督方法是指在训练过程中需要标注数据, 通过学习图像和标签之间的映射关系来实现分割. 早期的肝癌病理分割算法主要采用传统的图像处理方法, 如阈值分割、边缘检测、形态学处理等. 这些方法虽然简单易实现, 但精度有限, 难以应对复杂的肝癌图像. 深度学习技术的发展, 基于CNN的肝癌分割方法逐渐成为主流. 早期的深度学习模型主要是基于全连接层的神经网络, 如AlexNet[32]、VGG等. 这些模型的主要缺点是参数量大、容易过拟合. 随着研究的深入, U-Net[33]模型被提出, 图2是它的框架结构, 它采用编码器-解码器的结构, 编码器部分使用了卷积和池化操作来提取高层次特征, 解码器部分使用了反卷积和上采样操作来将特征映射回原始图像中, 而其中的跳跃连接可以处理医学图像中出现的小样本问题. 现有的U-Net框架主要用于先前的研究, 但并非所有由编码器模块提取的特征都适用病理图像分割. Zhai等人[34]以U-Net体系结构为主干, 在此基础上, 引入了注意引导连接模块(AGC). AGC模块的核心是注意力机制, 它可以自适应地选择有用的上下文特征, 同时抑制无用的特征, 以便更好地分割目标区域. 数据集方面, 将95个肝癌病理图像进行预处理, 获得了75562个可以训练的病理图像块, 其中训练集包含56672给图像补丁, 测试集包含18890个图像补丁, 通过在数据集上的比较, IoU增加了5.4%. Park等人[35]使用了一种改进的U-Net++模型, 该模型采用了深度监督的编码器-解码器网络结构. 在编码路径中, 使用了EfficientNet-B0[36]作为特征提取器, 该网络结构在保证高效计算的同时, 具有较强的特征提取能力, 框架示意图如图3所示. 为了进一步提高分割的准确性, 还采用了级联和卷积操作来获得最终结果. 在PAIP-2019 challenge数据集上的实验结果显示, 相较于传统U-Net模型, 这种改进的深度学习模型在肝癌分割任务中取得了更好的效果.

|
图 2 U-Net框架 |

|
图 3 U-Net++网络结构图 |
细胞核分割的一个主要挑战是分割可变大小的接触核. 由于可变大小的细胞核之间可能存在相互遮盖或者重叠的情况, 这会给细胞核的分割任务带来一定的困难. Rong等人[37]选用TME系统作为特征提取器, 使用YOLO模型进行细胞核分割, 模型提供的高质量的掩膜, 使得模型在分割细胞核时具有更高的准确性. 周志等人[38]提出了一种改进的U-Net模型, 使用深度特征聚合结构和高级监督的学习方法, 对细胞核进行精准分割. Lal等人[39]使用了一种强大的深度学习网络架构NucleiSegNet[40], 采用编码器-解码器结构, 架构中主要包含了一个鲁棒的残差块, 一个瓶颈块和一个注意解码器块. 该架构在提取和学习深度特征时可以去除不相关区域, 还解决了分割接触细胞核和准确检测具有可变形状的细胞核的挑战. 在KMC数据集上获得了97%的准确率.
大多病理图像中正常肝组织细胞和肿瘤细胞之间的差异很小, 而且具有相同类型的组织结构, U-Net的跳跃连接无法满足实验对特征信息的需求, 给分割的过程造成了很大的困难. Zhu等人[41]提出了一个融合多层次特征的模型, 采用多尺度扩张卷积神经网络模型对癌症病理图像进行分割. 模型中同样使用了和U-Net模型中相同的编码器-解码器网络, 将其设计为在网络中相同级别融合相同大小的特征图, 另外, 在其中增加了一个扩展卷积特征金字塔模块, 用于聚合多尺度信息. 不同内容、不同尺度的特征融合使整个模型能够有效应对目标尺度的变化, 在56672张可用于训练的病理图像中, 模型的Dice系数为 0.863. 同时, 与经典的网络模型相比, 该模型可以融合更多不同层次的图像信息.
研究人员选择U-Net作为改进基础, 很大程度上在于其跳跃连接可以结合低级和高级的特征使得在数据样本数量有限的情况下也能取得很好的性能, 打破了病理学数据较少的局限性. 这些改进的分割模型在U-Net编码器-解码器结构的基础上引入了一些新模块和新卷积或者更新了的特征提取和融合方法. 结果表明, 这些方法不仅提高了分割准确率, 还提高了模型的泛化能力, 具有很高的实用价值. 但需要注意的是模型性能提高的同时, 计算量也大幅度增加, 导致计算成本较大, 未来可以加强轻量化模型的研究. 还需要指出的是, 肝癌病理学图像分割问题是一个复杂的任务, 涉及到数据质量、标注精度等多个因素的影响, 在实验进行之前应当思考全面, 防止其他因素对实验结果产生影响.
3.3 术前预测肝切除术是肝肿瘤最常用的治疗方法. 然而, 术后高复发率严重影响这些HCC患者的长期预后[42].
但深度学习和数字WSI的结合在很大程度上减轻了实验的工作量, 揭示了不同实体瘤预后模型的良好准确性和有效性[43]. 对于制定肝切除术的手术方案、术后治疗和预后判断, 评估肝癌的恶性等级和判断微血管侵犯(MVI)具有重要意义.
3.3.1 病理分级肝细胞癌的病理分级是指根据肝癌细胞在组织学上的分化程度进行分类, 通常是根据不同的组织学特征来判断癌细胞的分化程度, 包括细胞形态、核大小和形态、核染色质形态、核仁数目和大小等方面. 在肝癌治疗中, 手术切除是一种常用的治疗方式. 对于早期的肝癌患者, 手术切除的治疗效果通常较好. 而对于晚期的肝癌患者, 则需要结合其他治疗方式, 如化疗、放疗、靶向治疗等. 因此, 在制定治疗方案时, 肝癌分级是一个重要的参考因素[44]. 一般来说, 肝癌的组织学分级越高, 预后越差, 治疗效果也越差[45]. 而且相对于低级别HCC患者, 大多数的高级别HCC病人在手术切除时需要更大的安全切缘, 以及更频繁的术后随访[46]. 在制定肝癌的治疗方案时, 需要结合患者的具体情况, 综合考虑肝癌的组织学等级, 制定个体化的治疗方案, 以达到最佳的治疗效果和预后. Aatresh等人[47]将肝细胞癌患者肝脏组织病理学数据分为4个级别, 即非癌、低亚型肝HCC肿瘤、中亚型肝HCC肿瘤和高亚型肝HCC肿瘤, 在LiverNet模型中加入注意力机制模块和萎缩空间金字塔池(ASPP)块, 结果显示, 该方法在KMC肝脏数据集上有90.93%的准确率, 在TCGA数据集上有97.72%的准确率. Chen等人[48]使用InceptionV3模型进行分级实验, 评估组织病理学分级(好、中、差)的MCC为0.738. 与3位分别有2年、5年和10年工作经验的病理学家相比, 分类器的性能几乎达到了有5年工作经验的病理学家的能力. 这表明卷积神经网络模型可以为肝癌治疗提供更为精准、可靠的肝癌分级信息, 在肝癌组织学分级方面具有很高的准确性和可靠性.
Edmondson和Steiner分级是确定HCC分级的常用标准[49], 它定义了以下4个肿瘤分级: G1、G2、G3和G4, 其中G1是最低分级, G4是最高分级. 图4[50]展示了正常组织G0和不同恶性等级的HCC组织学图像.
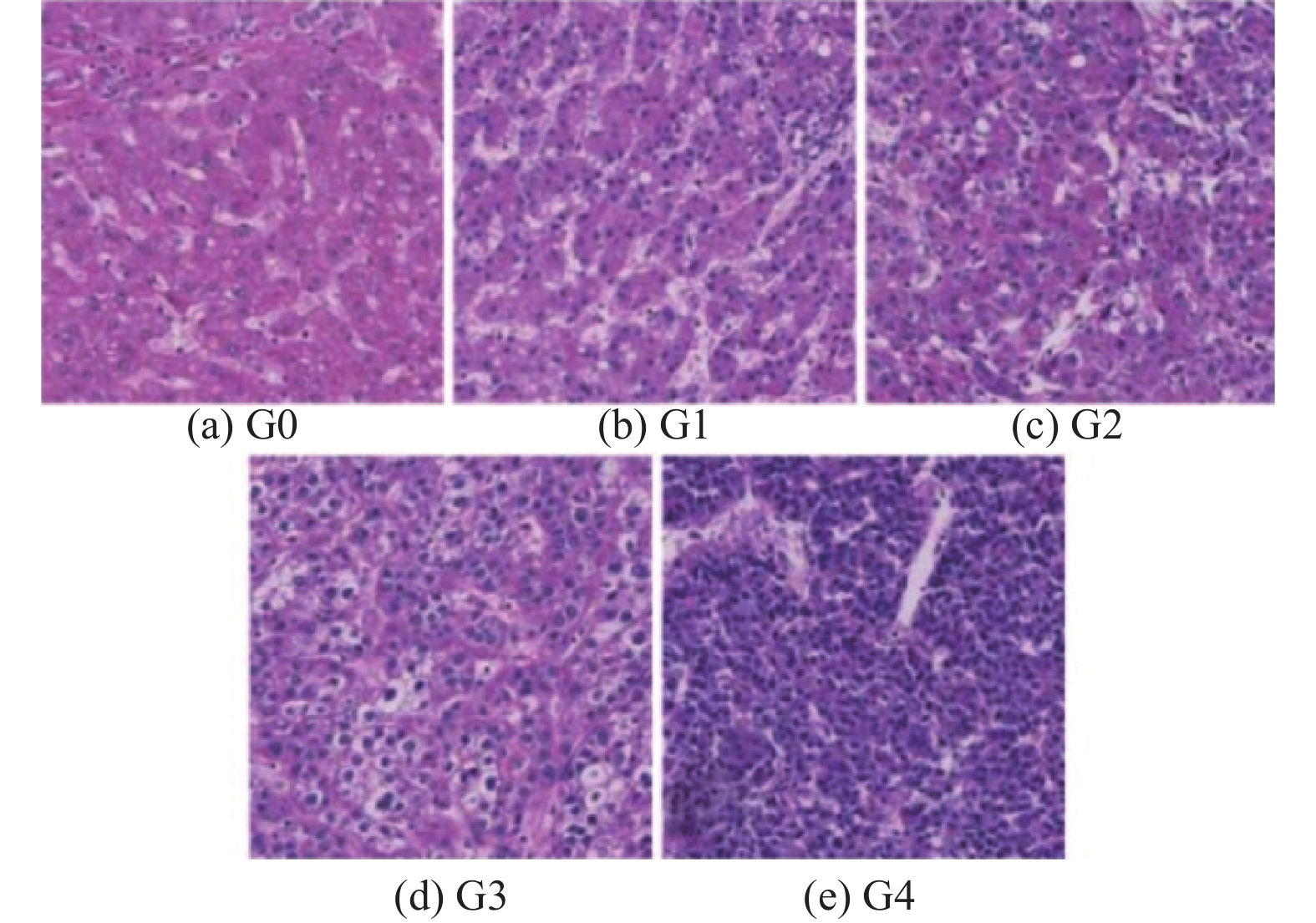
|
图 4 不同等级的组织图像 |
图像的特征是分级的关键, 研究人员为如何获取特征做出了很多努力, Li等人[51]利用多个完全连通的图层自动获得输入地图的多种形式的完整特征, 然后使用多重全连接卷积神经网络对细胞核进行自动分级. 使用某大型医院病理科3位病理学家在指导下获得的127张肝脏病理图像和相应标签作为实验数据, 结果表明, 使用的模型拥有较高的分级性能. Atupelage等人[50]通过计算获取到细胞核核内部的纹理、几何结构、空间分布特征. 进行分级时, 定义了一个管道路径, 通过核特征进行细胞核分级. 上述两种算法都是通过提取细胞核的特征进而实现分级任务的. 因为肝癌细胞核通常表现为细胞核变大、异形、核仁增大、染色质增多等特征. 这些形态学特征可以更好地反映肝癌细胞的分化程度和恶性程度, 从而得出更为准确的分级结果.
为了提高模型的性能, Chen等人[52]在一个拥有444张肝癌组织病理学图像的私有数据集上使用SENet[53]模型进行实验, 模型中的SE块可以挤压压缩模型结构中的每个特征层, 并使用激励来捕捉特征通道相关性. 另外, 模型中还加入了视觉注意力机制, 可以通过注意力可视化增加模型的解释形式. 在实验的结尾, 与VGG16、ResNet50[54]、ResNet_CBAM[55]和SKNet[56]这4种深度学习模型进行比较, SENet模型的准确率最高. 实验结果表明, SENet模型在图像分类任务中表现优秀, 并且具有较强的泛化能力, 这使得SENet模型成为当前深度学习领域中备受关注的模型之一. 研究人员进一步探究了SENet模型的内部机制, 发现SE块能够帮助模型自适应地学习输入数据的特征, 并且可以自适应地调整特征通道的权重. 这种自适应能力可以使得模型对输入数据的变化更加鲁棒[57]. Chanchal等人[58]使用一种包含空洞卷积(Atrous)思想的卷积神经网络模型来进行分级实验, 通过空洞卷积来学习具有大感受野的特征, 同时不牺牲空间分辨率. 模型来源于对BreastNet中ASPP块的改进, 并且, 实验尝试修改ASPP块的连接方式, 将密集连接和使用更少的串联, 同时通过以树状方式堆叠两种方式做对比, 实验结果表明, 在KMC肝脏病理图像数据集中的准确率分别为97.50%和97.14%. 实验表明, Atrous是一种高效的卷积操作, 可以在保证模型性能的同时, 降低计算复杂度和总的可训练参数.
多光子显微镜是一种用于成像生物样本的高分辨率显微镜技术, 它可以通过激光束的非线性光学效应, 对样本进行三维成像[59], 同时具有低光损伤和较深成像深度的优点. 与肝癌病理的结合提供了一种创新的计算机辅助诊断方法. Lin等人[60]将多光子显微镜用于肝癌病理, 然后和深度学习技术融合, 通过双光子激发荧光和二次谐波生成的组合图像训练基于VGG16框架的卷积神经网络, 该模型包括一个输入层、13个卷积层、3个全连接层和1个Softmax分类器层组成. 为了满足运算过程的高计算效率和低内存要求, 使用了初始学习率(lr)为0.0004的Adam优化算法来优化网络的权重和偏差; 同时, 选择交叉熵函数作为损失函数来衡量我们模型的性能, 在113名患者的切片图像中分化等级的分类准确率超过90%. 无标签、自动化方法在医学图像处理领域具有很大的应用潜力, 可以提高医学图像处理的效率和准确性, 同时也可以减少人工标注数据的工作量. 然而, 由于医学图像数据的复杂性和多样性, 这些方法在实际应用中仍存在一些挑战和难点, 例如模型的稳定性、泛化能力、数据量不足等问题. 因此, 需要进一步加强算法的研究和改进, 以实现更为准确和可靠的医学图像处理.
上述实验介绍了在病理学图像中卷积神经网络在肝癌分级实验中的一些研究进展. 总体而言, 该实验是一个具有挑战性的任务, 过程中需要考虑到多方面可能出现的问题. 肝癌分级在肝癌治疗中具有重要的参考价值, 可以帮助医生制定合适的治疗方案, 提高治疗效果.
3.3.2 微血管侵犯微血管侵犯(MVI)指的是肝癌细胞侵入肝脏的微血管, 从而导致肿瘤在肝脏内快速扩散和生长的现象, 是癌症术后长期生存率低的主要原因. MVI可以反映出肝癌的恶性程度, 早期发现和治疗对于提高生存率非常重要. 而MVI的识别是非常具有挑战性的, 因为大多数MVI样本太小, 并且由新的区别特征组成, 无疑给实验带来了极大的挑战.
Feng等人[61]在检测MVI时采用ResNet18作为模型的编码器, 主要思想是通过使用斑块筛选和动态标签平滑策略, 从输入和输出的角度处理带有噪声注释的肝脏组织病理学图像, 降低噪声负面影响, 提高模型效果. Chen等人[62]在多中心HCC队列中开发了MVI深度学习预测模型, 该框架由CNN特征提取层、弱监督多实例学习(MIL)池化层和全连接层组成. 通过学习组织学WSI肿瘤区域的特征信息, 自动准确地评估MVI状态, 使用TCGA数据集中的376个WSI图像进行实验, 在测试集上的AUC达到了90.4%. Sun等人[63]使用某外科医院提供的69个肝脏组织病理学图像的WSI数据集进行实验, 根据MVI边界组织病理学结构的特点, 将卷积神经网络(CNN)方法与视觉变换器相结合, 引入视觉转换器可以有效地提取所需的全局特征, 精确度和F1分数高于ResNet101、InceptionV3. 补充了CNN网络的优势, 提高了MVI边界图像的识别性能, 在数据集上的F1分数达到80.06%. 为了进一步提高实验的效率, Chen等人[64]使用级联网络的三阶段系统来检测HCC组织WSI上的MVI. 区域特征提取和分割分别采用了SE-ResNet和SE-ResNet-U-Net模型, SE-ResNet的优点在于它允许基于其全局平均值对特征图进行自调整, 可以更加适合模型的训练和优化. 考虑到大多数现有的计算病理学算法在很大程度上忽略了将可疑组织与其周围的邻居进行比较, 在检测MVI时, 采用图卷积神经网络(GCN)进行分类. 实验的F1分数为87.8%. 以上研究仅预测了MVI是否存在, 并没有进一步对MVI做出更细致的分类. Wang等人[65]尝试通过分析特征将患者分为3期: M0 (无MVI)、M1 (低风险)和M2 (高风险), 然后通过构建2层前馈神经网络, 建立深度学习预后预测模型. 网络整体使用梯度下降进行优化. 实验结果显示, 该方法与传统的分期系统相比有更好的预后预测, 可以对复发风险较高的患者进行更个性化的管理. 但是实验中根据特征的分析得到的分期标准是通过病理学家的讨论研究得出的, 未被广泛接受.
研究人员利用卷积神经网络开发出多种MVI检测模型, 能够自动准确地评估MVI状态. 这些模型的应用可以帮助医生更好地评估肝癌患者的病情和手术预后, 并为其提供更加个性化的治疗方案. 对于肝癌的诊断和治疗来说, 检测MVI具有重要的临床意义.
3.4 小结基于CNN的方法是肝癌诊断中最常用的方法, 也是目前深度学习中应用最广泛的方法, CNN应用于肝肿瘤检测, 图像分割和术前预测有助于提高肝癌的早期诊断和治疗效果, 为医生提供了更准确和可靠的辅助工具. 基于CNN的肝癌病理学图像诊断算法比较如表2所示, 基于CNN的肝癌病理学图像诊断算法的改进及效果如表3所示.
| 表 2 基于CNN的肝癌病理学图像诊断算法比较 |
| 表 3 基于CNN的肝癌病理学图像诊断算法的改进及效果 |
4 总结与展望
在这个医疗科技创新时代, 深度学习技术中的CNN模型凭借对医学图像的高效和准确的诊断表现, 已经成为越来越多研究人员所偏爱的实验模型. 肝癌组织病理学图像相比于影像学图像可以观察到肿瘤排列方式等详细信息, 而且在治疗规划时具有较高的精确性和个性化程度, 对于诊断的准确性和治疗效果的提高都有重要意义. 与传统的机器学习算法相比, 在肝癌病理学诊断中CNN的优点如下.
(1) CNN可以自动从病理学图像中学习特征, 而无需手动设计特征. 这种自动特征提取的能力可以减轻医生的工作负担, 提高病理学图像的诊断准确性.
(2)病理学图像通常具有复杂的结构和纹理, 同时还会受到光线、噪声等因素的干扰. 相比传统机器学习算法, CNN具有更强的鲁棒性, 可以在这些复杂情况下仍能提取出有效的特征.
(3)病理学图像通常包含大量的像素, 且数据量庞大. 相比传统机器学习算法, CNN可以通过高度并行化的计算方式, 快速处理大量数据, 并在较短的时间内完成诊断.
(4) CNN的模型参数可以在不同的病理学图像数据集之间共享. 这种可迁移性使得CNN可以在不同数据集上进行泛化, 从而提高模型的泛化能力.
卷积神经网络在肝癌病理学诊断中的应用已经取得了很大的进展, 但仍存在一些挑战和缺点.
(1)由于CNN模型通常需要大量的参数, 因此它们容易发生过度拟合, 特别是在小样本训练集的情况下. (2)虽然CNN在图像分类任务中表现出色, 但它们的决策过程往往是不可解释的.
卷积神经网络在病理学图像诊断方面的应用相对较新, 在近几年才开始得到广泛的应用. 由于数据样本本身的局限性, 在进行实验之前首先要考虑样本问题, 才能对模型的性能进行改进. 在进行实验之前, 可以根据面临的问题选择一些适合小样本的模型, 或者在实验中采取数据增强、迁移学习或者改变损失函数等方式. 而对于CNN的改进基本可以通过引入新模块和引入新卷积来实现.
(1)在卷积神经网络中引入残差块, 可以帮助解决梯度消失问题, 从而加速训练和提高模型性能.
(2)使用注意力机制模块来自适应地调整不同卷积层中的特征图的重要性, 从而提高模型的性能.
(3)反卷积是一种可以将低分辨率特征图上采样到高分辨率特征图的技术. 在CNN中, 反卷积可以用于图像分割和目标检测等任务中.
(4)空洞卷积是一种可以增加感受野(一个特定的输出神经元对输入数据的局部区域感知的范围)大小的技术, 可以通过在卷积核中引入空洞来扩大卷积核的有效感受野. 同时, 还有其他一些模块和技术, 如卷积核的形状、池化方式、Dropout等, 也可以用于改进CNN.
CNN模型在肝癌病理学图像诊断方面的应用已经取得了巨大的进展, 但仍然存在一些问题, 因此在未来的研究中可开展如下几个方面的工作.
(1)肝癌病理学图像数据较少, 很多实验者会采取一些方法进行扩展训练集, 这种方式不免会使计算量增大, 因此未来的研究可以探索如何将CNN模型更好地应用于肝癌病理学图像的诊断中, 在大幅增加计算量的同时还可以提高病理学图像诊断的准确性和效率.
(2)对于肝癌病理诊断任务, 不同医院和实验室采集的数据可能存在一定的差异, 可能会影响模型的泛化能力. 迁移学习可以通过利用已有的模型在其他领域训练的经验, 提高模型在新数据集上的效果. 因此, 未来应将更多的研究放在探索如何利用迁移学习来提高肝癌病理诊断的准确性和可靠性上.
(3)卷积神经网络是黑盒模型, 难以解释其预测结果的原因. 在医疗领域, 模型的可解释性很重要, 因为医生需要了解模型的判断依据, 从而更好地理解和使用模型的结果. 未来会有更多的研究探索如何将可解释性深度学习方法应用到肝癌病理诊断任务中, 提高模型的可解释性和可靠性.
(4)除了病理图像数据, 医学影像数据、基因数据等多种数据模态也可以提供对肝癌的诊断和治疗的重要信息. 因此, 将多个数据模态融合起来, 建立多模态深度学习模型, 可以进一步提高肝癌病理诊断的准确性和可靠性.
(5)实验过程中多种病理学变体、光照和噪声变化等问题. 在实际应用中, 未来应考虑多种数据增强方法的结合, 以实现最佳的数据增强效果.
(6)在分割实验中, 使用像素级标签进行分割难免会遇到类不平衡问题, 未来可以尝试在使用图像级标签的情况下, 还可以保证足够的细节和精确度来准确标注肝肿瘤的边界和形状.
5 结束语近年来, 深度学习在医疗诊断领域取得了显著的进展, 特别是卷积神经网络(CNN)在医学病理图像处理和分析方面的应用已经取得了重大的突破. 随着研究的深入, 肝癌自动诊断技术将进一步完善, 以提高临床对肝肿瘤病变早期侦查和治疗的效率.
| [1] |
Llovet JM, Pinyol R, Kelley RK, et al. Molecular pathogenesis and systemic therapies for hepatocellular carcinoma. Nature Cancer, 2022, 3(4): 386-401. DOI:10.1038/s43018-022-00357-2 |
| [2] |
Chen WQ, Zheng RS, Baade PD, et al. Cancer statistics in China, 2015. CA: A Cancer Journal for Clinicians, 2016, 66(2): 115-132. DOI:10.3322/caac.21338 |
| [3] |
van der Laak J, Litjens G, Ciompi F. Deep learning in histopathology: The path to the clinic. Nature Medicine, 2021, 27(5): 775-784. DOI:10.1038/s41591-021-01343-4 |
| [4] |
LeCun Y, Bengio Y, Hinton G. Deep learning. Nature, 2015, 521(7553): 436-444. DOI:10.1038/nature14539 |
| [5] |
Lee SH, Chan CS, Mayo SJ, et al. How deep learning extracts and learns leaf features for plant classification. Pattern Recognition, 2017, 71: 1-13. DOI:10.1016/j.patcog.2017.05.015 |
| [6] |
LeCun Y, Boser B, Denker J, et al. Handwritten digit recognition with a back-propagation network. Advances in Neural Information Processing Systems, 1989, 2.
|
| [7] |
Azer SA. Deep learning with convolutional neural networks for identification of liver masses and hepatocellular carcinoma: A systematic review. World Journal of Gastrointestinal Oncology, 2019, 11(12): 1218-1230. DOI:10.4251/wjgo.v11.i12.1218 |
| [8] |
江宇, 柳宗翰, 程树群. 人工智能在肝癌领域应用研究进展. 中国实用外科杂志, 2022, 42(12): 1428-1431. DOI:10.19538/j.cjps.issn1005-2208.2022.12.16 |
| [9] |
陈钇地, 蒋涵羽, 陈婕, 等. 肝癌影像学机遇与挑战——中国十年来发展成果及展望. 磁共振成像, 2022, 13(10): 71-78. |
| [10] |
Feng B, Ma XH, Wang S, et al. Application of artificial intelligence in preoperative imaging of hepatocellular carcinoma: Current status and future perspectives. World Journal of Gastroenterology, 2021, 27(32): 5341-5350. DOI:10.3748/wjg.v27.i32.5341 |
| [11] |
刘文广, 谢斯敏, 周雅芳, 等. 机器学习及其在肝脏疾病影像诊断中的研究进展. 国际医学放射学杂志, 2019, 42(1): 16-21. DOI:10.19300/j.2019.z6102zt |
| [12] |
Gul S, Khan MS, Bibi A, et al. Deep learning techniques for liver and liver tumor segmentation: A review. Computers in Biology and Medicine, 2022, 147: 105620. DOI:10.1016/j.compbiomed.2022.105620 |
| [13] |
Schneider L, Laiouar-Pedari S, Kuntz S, et al. Integration of deep learning-based image analysis and genomic data in cancer pathology: A systematic review. European Journal of Cancer, 2022, 160: 80-91. DOI:10.1016/j.ejca.2021.10.007 |
| [14] |
Kim YJ, Jang H, Lee K, et al. PAIP 2019: Liver cancer segmentation challenge. Medical Image Analysis, 2021, 67: 101854. DOI:10.1016/j.media.2020.101854 |
| [15] |
Tomczak K, Czerwińska P, Wiznerowicz M. The Cancer Genome Atlas (TCGA): An immeasurable source of knowledge. Contemporary Oncology/Współczesna Onkologia, 2015, 19(1A): A68-A77. |
| [16] |
Singh M, Singh S, Gupta S. An information fusion based method for liver classification using texture analysis of ultrasound images. Information Fusion, 2014, 19: 91-96. DOI:10.1016/j.inffus.2013.05.007 |
| [17] |
Das A, Das P, Panda SS, et al. Detection of liver cancer using modified fuzzy clustering and decision tree classifier in CT images. Pattern Recognition and Image Analysis, 2019, 29(2): 201-211. DOI:10.1134/S1054661819020056 |
| [18] |
Qiu W, Xiao F, Yang X, et al. Research on fuzzy enhancement in the diagnosis of liver tumor from b-mode ultrasound images. International Journal of Image, Graphics and Signal Processing, 2011, 3(3): 10-16. DOI:10.5815/ijigsp.2011.03.02 |
| [19] |
Vapnik VN. The Nature of Statistical Learning Theory. 2nd ed., New York: Springer, 1999.
|
| [20] |
Tanwar N, Rahman KF. Machine learning in liver disease diagnosis: Current progress and future opportunities. IOP Conference Series: Materials Science and Engineering, 2021, 1022: 012029. DOI:10.1088/1757-899X/1022/1/012029 |
| [21] |
宋杰, 肖亮, 练智超, 等. 基于深度学习的数字病理图像分割综述与展望. 软件学报, 2021, 32(5): 1427-1460. DOI:10.13328/j.cnki.jos.006205 |
| [22] |
Saba T. Recent advancement in cancer detection using machine learning: Systematic survey of decades, comparisons and challenges. Journal of Infection and Public Health, 2020, 13(9): 1274-1289. DOI:10.1016/j.jiph.2020.06.033 |
| [23] |
宋国利, 陈杰. 病理图像分析的深度学习方法研究综述. 中国科学基金, 2022, 36(2): 225-234. DOI:10.16262/j.cnki.1000-8217.2022.02.033 |
| [24] |
Huang WC, Chung PC, Tsai HW, et al. Automatic HCC detection using convolutional network with multi-magnification input images. Proceedings of the 2019 IEEE International Conference on Artificial Intelligence Circuits and Systems (AICAS). Hsinchu: IEEE, 2019. 194–198.
|
| [25] |
Simonyan K, Zisserman A. Very deep convolutional networks for large-scale image recognition. Proceedings of the 3rd International Conference on Learning Representations. San Diego: ICLR, 2015.
|
| [26] |
Yang TL, Tsai HW, Huang WC, et al. Pathologic liver tumor detection using feature aligned multi-scale convolutional network. Artificial Intelligence in Medicine, 2022, 125: 102244. DOI:10.1016/j.artmed.2022.102244 |
| [27] |
Diao SH, Luo WR, Hou JX, et al. Computer aided cancer regions detection of hepatocellular carcinoma in whole-slide pathological images based on deep learning. Proceedings of the 2019 International Conference on Medical Imaging Physics and Engineering (ICMIPE). Shenzhen: IEEE, 2019. 1–6.
|
| [28] |
Hou L, Samaras D, Kurc TM, et al. Patch-based convolutional neural network for whole slide tissue image classification. Proceedings of the 2016 IEEE Conference on Computer Vision and Pattern Recognition. Las Vegas: IEEE, 2016. 2424–2433.
|
| [29] |
Wang J, Xu Z, Pang ZF, et al. Tumor detection for whole slide image of liver based on patch-based convolutional neural network. Multimedia Tools and Applications, 2021, 80(11): 17429-17440. DOI:10.1007/s11042-020-09282-x |
| [30] |
Nguyen C, Asad Z, Deng R, et al. Evaluating transformer-based semantic segmentation networks for pathological image segmentation. Medical Imaging 2022: Image Processing. SPIE, 2022. 942–947.
|
| [31] |
Roy S, Das D, Lal S, et al. Novel edge detection method for nuclei segmentation of liver cancer histopathology images. Journal of Ambient Intelligence and Humanized Computing, 2023, 14(1): 479-496. DOI:10.1007/s12652-021-03308-4 |
| [32] |
Krizhevsky A, Sutskever I, Hinton GE. ImageNet classification with deep convolutional neural networks. Communications of the ACM, 2017, 60(6): 84-90. DOI:10.1145/3065386 |
| [33] |
Ronneberger O, Fischer P, Brox T. U-Net: Convolutional networks for biomedical image segmentation. Proceedings of the 18th International Conference on Medical Image Computing and Computer-assisted Intervention. Munich: Springer, 2015. 234–241.
|
| [34] |
Zhai ZR, Wang CL, Sun ZQ, et al. Deep neural network guided by attention mechanism for segmentation of liver pathology image. Proceedings of the 2021 Chinese Intelligent Systems Conference, Volume III. Singapore: Springer, 2022. 425–435.
|
| [35] |
Park HC, Ghimire R, Poudel S, et al. Deep learning for joint classification and segmentation of histopathology image. Journal of Internet Technology, 2022, 23(4): 903-910. DOI:10.53106/160792642022072304025 |
| [36] |
Tan MX, Le QV. EfficientNet: Rethinking model scaling for convolutional neural networks. Proceedings of the 36th International Conference on Machine Learning. Long Beach: ICML, 2019. 6105–6114.
|
| [37] |
Rong RC, Sheng HDY, Jin KW, et al. A deep learning approach for histology-based nucleus segmentation and tumor microenvironment characterization. Modern Pathology, 2023, 36(8): 100196. DOI:10.1016/j.modpat.2023.100196 |
| [38] |
周志, 张孙杰, 张晓玥. 一种改进U型神经网络的医学细胞核图像分割方法. 小型微型计算机系统, 2023, 44(1): 110-116. |
| [39] |
Lai S, Das D, Alabhya K, et al. NucleiSegNet: Robust deep learning architecture for the nuclei segmentation of liver cancer histopathology images. Computers in Biology and Medicine, 2021, 128: 104075. DOI:10.1016/j.compbiomed.2020.104075 |
| [40] |
Metaxas D, Qu H, Riedlinger G, et al. Deep learning-based nuclei segmentation and classification in histopathology images with application to imaging genomics. Computer Vision for Microscopy Image Analysis. Amsterdam: Elsevier, 2021. 185–201.
|
| [41] |
Zhu P, Wang CL, Sun ZQ, et al. Segmentation of liver cancer pathology images based on multi-scale feature fusion. Proceedings of the 2021 Chinese Intelligent Systems Conference: Volume III. Singapore: Springer, 2022. 596–605.
|
| [42] |
林鹏, 蔡敏清, 房俊伟, 等. 肝癌术后复发的外科治疗研究现状及进展. 器官移植, 2022, 13(1): 111-119. |
| [43] |
Qu WF, Tian MX, Qiu JT, et al. Exploring pathological signatures for predicting the recurrence of early-stage hepatocellular carcinoma based on deep learning. Frontiers in Oncology, 2022, 12: 968202. DOI:10.3389/fonc.2022.968202 |
| [44] |
Deuffic S, Poynard T, Buffat L, et al. Trends in primary liver cancer. The Lancet, 1998, 351(9097): 214-215. |
| [45] |
Marrero JA, Kulik LM, Sirlin CB, et al. Diagnosis, staging, and management of hepatocellular carcinoma: 2018 practice guidance by the american association for the study of liver diseases. Hepatology, 2018, 68(2): 723-750. DOI:10.1002/hep.29913 |
| [46] |
韩玉齐, 魏靖伟, 蒋涵羽, 等. 影像组学研究进展及其在肝癌中的临床应用. 中国科学: 生命科学, 2020, 50(11): 1309-1320. |
| [47] |
Aatresh AA, Alabhya K, Lal S, et al. LiverNet: Efficient and robust deep learning model for automatic diagnosis of sub-types of liver hepatocellular carcinoma cancer from H&E stained liver histopathology images. International Journal of Computer Assisted Radiology and Surgery, 2021, 16(9): 1549-1563. DOI:10.1007/s11548-021-02410-4 |
| [48] |
Chen MY, Zhang B, Topatana W, et al. Classification and mutation prediction based on histopathology H&E images in liver cancer using deep learning. NPJ Precision Oncology, 2020, 4(1): 14. DOI:10.1038/s41698-020-0120-3 |
| [49] |
Edmondson HA, Steiner PE. Primary carcinoma of the liver. A study of 100 cases among 48900 necropsies. Cancer, 1954, 7(3): 462-503. |
| [50] |
Atupelage C, Nagahashi H, Kimura F, et al. Computational hepatocellular carcinoma tumor grading based on cell nuclei classification. Journal of Medical Imaging, 2014, 1(3): 034501. DOI:10.1117/1.JMI.1.3.034501 |
| [51] |
Li SQ, Jiang HY, Pang WB. Joint multiple fully connected convolutional neural network with extreme learning machine for hepatocellular carcinoma nuclei grading. Computers in Biology and Medicine, 2017, 84: 156-167. DOI:10.1016/j.compbiomed.2017.03.017 |
| [52] |
Chen C, Chen C, Ma MR, et al. Classification of multi-differentiated liver cancer pathological images based on deep learning attention mechanism. BMC Medical Informatics and Decision Making, 2022, 22(1): 176. DOI:10.1186/s12911-022-01919-1 |
| [53] |
Hu J, Shen L, Sun G. Squeeze-and-excitation networks. Proceedings of the 2018 IEEE/CVF Conference on Computer Vision and Pattern Recognition. Salt Lake City: IEEE, 2018. 7132–7141.
|
| [54] |
He KM, Zhang XY, Ren SQ, et al. Deep residual learning for image recognition. Proceedings of the 2016 IEEE Conference on Computer Vision and Pattern Recognition. Las Vegas: IEEE, 2016. 770–778.
|
| [55] |
Woo S, Park J, Lee JY, et al. CBAM: Convolutional block attention module. Proceedings of the 15th European Conference on Computer Vision (ECCV). Munich: Springer, 2018. 3–19.
|
| [56] |
Li X, Wang WH, Hu XL, et al. Selective kernel networks. Proceedings of the 2019 IEEE/CVF Conference on Computer Vision and Pattern Recognition. Long Beach: IEEE, 2019. 510–519.
|
| [57] |
Li X, Shen X, Zhou YX, et al. Classification of breast cancer histopathological images using interleaved DenseNet with SENet (IDSNet). PLoS One, 2020, 15(5): e0232127. DOI:10.1371/journal.pone.0232127 |
| [58] |
Chanchal AK, Lal S, Barnwal D, et al. Evolution of livernet 2.x: Architectures for automated liver cancer grade classification from H&E stained liver histopathological images. Multimedia Tools and Applications, 2023: 1–31. [doi: 10.1007/s11042-023-15176-5]
|
| [59] |
Dunn KW, Young PA. Principles of multiphoton microscopy. Nephron Experimental Nephrology, 2006, 103(2): e33-e40. DOI:10.1159/000090614 |
| [60] |
Lin HX, Wei C, Wang GX, et al. Automated classification of hepatocellular carcinoma differentiation using multiphoton microscopy and deep learning. Journal of Biophotonics, 2019, 12(7): e201800435. DOI:10.1002/jbio.201800435 |
| [61] |
Feng S, Yu XT, Liang WJ, et al. Development of a deep learning model to assist with diagnosis of hepatocellular carcinoma. Frontiers in Oncology, 2021, 11: 762733. DOI:10.3389/fonc.2021.762733 |
| [62] |
Chen QF, Xiao H, Gu YQ, et al. Deep learning for evaluation of microvascular invasion in hepatocellular carcinoma from tumor areas of histology images. Hepatology International, 2022, 16(3): 590-602. DOI:10.1007/s12072-022-10323-w |
| [63] |
Sun L, Sun ZQ, Wang CL, et al. PCformer: An MVI recognition method via classification of the MVI boundary according to histopathological images of liver cancer. Journal of the Optical Society of America A, 2022, 39(9): 1673-1681. DOI:10.1364/JOSAA.463439 |
| [64] |
Chen HB, Wang K, Zhu YY, et al. From pixel to whole slide: Automatic detection of microvascular invasion in hepatocellular carcinoma on histopathological image via cascaded networks. Proceedings of the 24th International Conference on Medical Image Computing and Computer Assisted Intervention. Strasbourg: Springer, 2021. 196–205.
|
| [65] |
Wang K, Xiang YJ, Yan JP, et al. A deep learning model with incorporation of microvascular invasion area as a factor in predicting prognosis of hepatocellular carcinoma after R0 hepatectomy. Hepatology International, 2022, 16(5): 1188-1198. DOI:10.1007/s12072-022-10393-w |
 2024, Vol. 33
2024, Vol. 33


